LinkRdata, une nouvelle visualisation des données pour les SHS : application à la cognition
#VALORISATION
Le partage des données est une pratique qui a révolutionné la biologie et les sciences de la santé en les faisant progresser et en réduisant les coûts. Il y a quelques années (2013-2014), je me suis intéressée à l’une des grandes plateformes de partage de données, la plateforme « 1000 connectome » qui regroupe des images par résonance magnétique fonctionnelle (IRMf) de 1 000 individus à travers le monde. J’ai réutilisé ces données et j’ai pu révéler l’existence de nouvelles régions du cerveau qui se co-activent ensemble au repos. Afin de compléter les résultats, j’ai cherché à créer un atlas fonctionnel pour le cerveau qui relie régions et fonctions.
La puissance de calcul des serveurs, les traitements informatiques et mathématiques ont simplifié ce travail : j'ai ainsi pu collecter des données, les stocker et faciliter leur accès. En revanche, j'ai eu des difficultés à nommer des fonctions cognitives ou sensorimotrices pour chacune des régions cérébrales que j'avais distinguées.
Le principal problème que pose le traitement des données est la multiplicité des échelles auxquelles elles sont exprimées. Par exemple, pour interpréter des images issues de l’imagerie de diffusion, le chercheur/clinicien a besoin d’autres types de données provenant de deux grands domaines scientifiques qui progressent à des rythmes différents :
- les données d’imagerie du cerveau humain liées à la cognition, dans des contextes sains et cliniques ;
- les études génomiques et protéomiques, obtenues à partir de modèles animaux et humains.
Pour relever le défi de l’intégration de données cérébrales multi-sources et multi-échelles, j'ai développé une première application dans le cadre de l’équipement d’excellence transdisciplinaire sur la mémoire, l’Equipex MATRICE. Les logiciels que je propose sous le nom de LinkRdata sont d’autant plus utiles à ce type de programmes (portés par l’InSHS) que leur caractère transdisciplinaire complexifie les bases de données produites et implique des instruments adaptés. Ainsi, LinkRdata est la continuation logique d’une série de projets scientifiques qui ont eu pour but de fédérer et d’intégrer un ensemble de bases de données appartenant à des disciplines différentes afin de créer de la connaissance autour de ces données.
La plateforme LinkRdata est basée uniquement sur des sources de données ouvertes. Ces données ont permis de construire des bases pour :
- des tâches cognitives et des pathologies du cerveau, en se basant sur 14 000 articles neurocognitifs ;
- les taux d’expressions d’environ 21 000 gènes dans 947 échantillons du cerveau ;
- les coordonnées et densités de fibres qui relient 1 015 régions du cerveau.
LinkRdata intègre ces connaissances cognitives, sensorimotrices, anatomiques et transcriptomiques, via trois logiciels : LinkRbrain, LinkRbiblio et LinkRfiber.
Les logiciels libres de LinkRdata
LinkRbrain
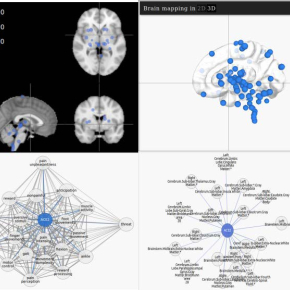
L’outil LinkRbrain — d’ores et déjà accessible à l’ensemble de la communauté par le biais d’un site web ouvert — a été développé afin de relever le défi d’intégrer les connaissances cognitives, pathologiques, génétiques et anatomiques sur les fonctions cérébrales. La plateforme développée accumule l’information de plusieurs bases de données et intègre ces données multi-échelles dans un cadre commun afin que chaque point du cerveau soit caractérisé par un profil cognitif, un profil d’expression génétique et un marqueur neuro-anatomique. Ainsi, LinkRbrain lie systématiquement :
- un ensemble de pics d’activation du cerveau vers un ensemble de marqueurs cognitifs ;
- un profil d’expression génétique vers un ensemble de marqueurs cognitifs ;
- un ensemble de marqueurs cognitifs ou profil d’expression génétique vers des marqueurs neuro-anatomiques.
LinkRbiblio
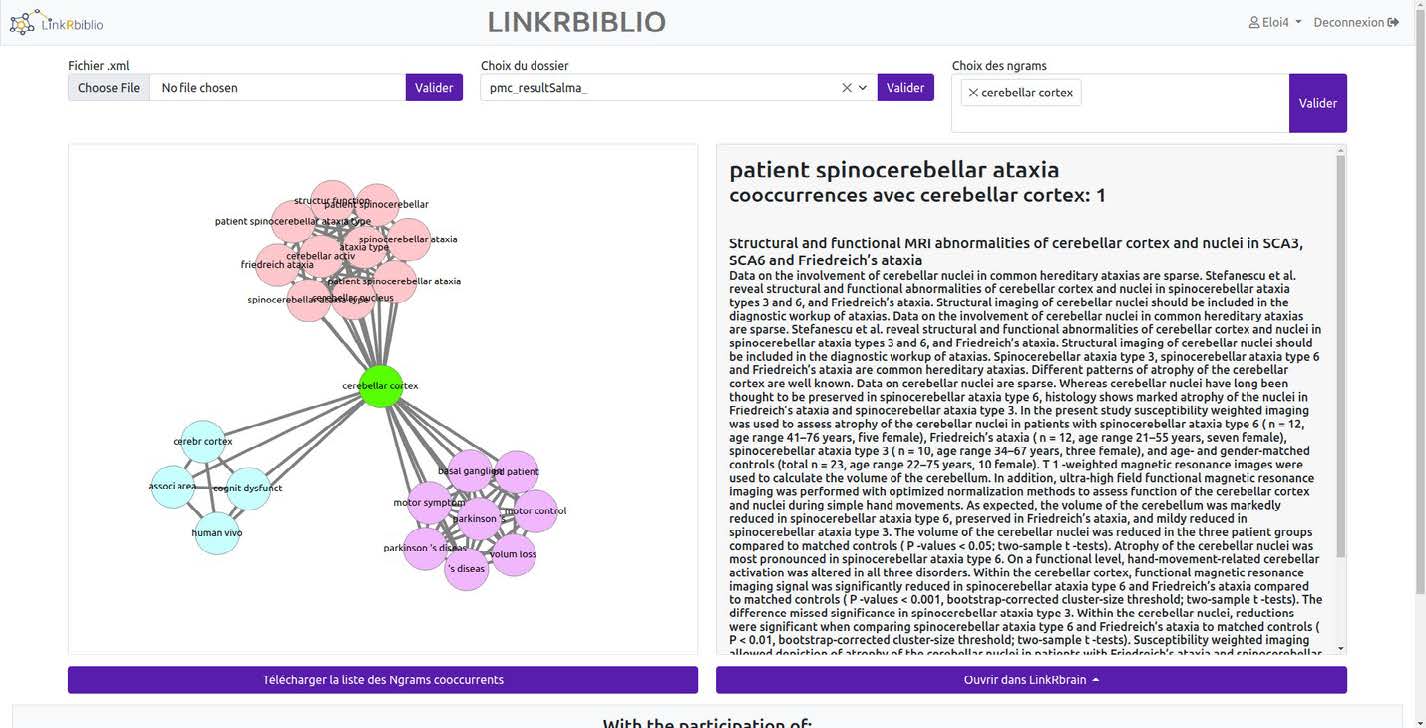
LinkRbiblio permet d’explorer les bases de données bibliographiques. LinkRbiblio extrait les n-grams (mots et séquences de mots) à partir du corpus constitué par les résumés ou les corps de textes et les titres de publications autour d’une thématique précise. Ce module du traitement du langage naturel attribue des étiquettes grammaticales à chaque mot, puis une annotation grammaticale est effectuée pour extraire les phrases nominales appropriées. Le choix de n-grams au lieu de mots uniques est crucial pour notre projet car le nom de certaines fonctions cognitives comme « visual working memory » est constitué de trois mots. La coprésence de deux n-grams est calculée à partir du nombre de publications qui contiennent ces deux n-grams. Cette valeur de la coprésence traduit la proximité entre les termes/n-grams utilisés par les auteurs. On distinguera la coprésence de la cooccurrence qui, elle, s’appuie sur un calcul de probabilité.
LinkRbiblio est utilisé aussi bien pour l’extraction des connaissances à partir du portail Pubmed (site qui indexe les papiers dans le domaine du biomédical) que pour un corpus en sciences humaines et sociales. Il peut déjà encourager des interactions entre historiennes, sociologues et psychologues. Grâce au développement d’une interface de programmation d’application (API) qui permet de relier LinkRbrain à LinkRbiblio, les n-grams extraits par textmining sont connectés aux activations du cerveau.
LinkRfiber
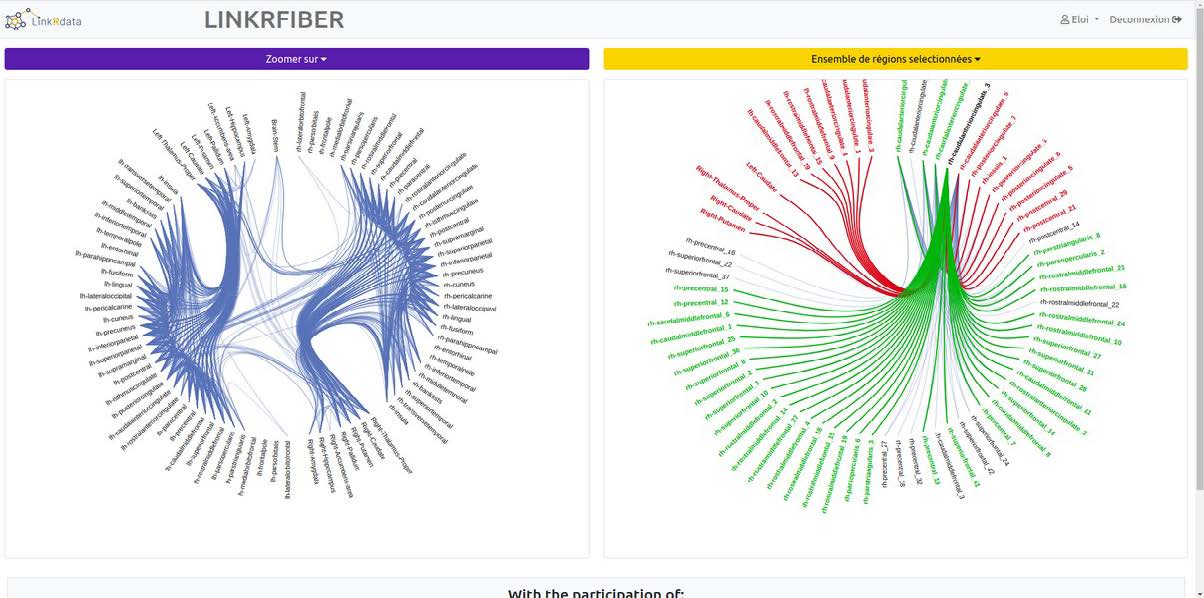
L’outil LinkRfiber permet de visualiser les connexions structurelles (fibres) entre 1 000 régions du cerveau à l’échelle individuelle. Pour alimenter en données notre outil, une base de données structurales a été récupérée à partir du projet Connectome. Ces données permettent de localiser les régions qui sont reliées par les fibres. Pour intégrer de la connaissance autour de ces données structurelles individuelles, j’ai utilisé l’API linkrbrain-client. Grâce à cette API, l’information structurale est reliée aux données cognitives, pathologiques et transcriptomiques.
Apports de la plateforme LinkRdata
La plateforme linkRdata permettra aux chercheurs et chercheuses de :
- Accélérer la fouille bibliographique et son appréhension synthétique visuelle.
- Éviter de fausses inférences par manque de connaissance : certaines fonctions cognitives sont largement connectées et il est impossible de connaître toute la bibliographie existante pour se faire une idée exhaustive de ces interactions. L’outil est donc utile pour, d’une part, préparer le design expérimental et, d’autre part, interpréter les données.
- Décloisonner les disciplines : les chercheurs et chercheuses sont experts d’un domaine. Il est très difficile de connaître toutes les interactions entre les différents systèmes quand ce ne sont pas celles que nous cherchons.
- Confronter ses résultats à la bibliographie.
- Associer la bibliographie à une agrégation de données de génomique : cette étape se fait d’ordinaire à la main ; elle représente une perte de temps et s’effectue au risque que l’objectivité fasse défaut.
La plateforme linkRdata aidera également les professionnelles de santé à identifier les réseaux cérébraux du patient qui pourraient être affectés par la mort neuronale, à les rendre ainsi plus confiants dans leurs décisions et à mieux orienter le traitement. Elle leur permettra de prédire les fonctions cognitives et sensorimotrices afin de mieux concevoir leurs études et accélérer les essais cliniques pour améliorer la plasticité du cerveau. Elle favorisera une meilleure gestion clinique des patients sujets à des handicaps suite à des atteintes cognitives et sensorimotrices et optimisera les dépenses du système de santé, en évitant ou en réduisant certains handicaps potentiels. Elle offrira enfin aux patients une connaissance approfondie des risques individuels et permettra une meilleure prise en charge, visant à améliorer leur bien-être. Ceux-ci auront également la possibilité d’enrichir la base de données et donc les connaissances sur leur pathologie.
Cas d’utilisation de LinkRdata
Visualisation de la littérature et son ordonnancement
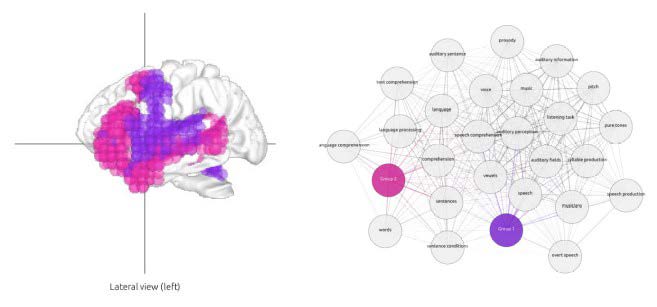
Les fonctions cognitives « parole » et « phrases » sont considérées comme deux fonctions distinctes. La fonction « parole » est liée à la production de mots, de syllabes et/ou de voyelles, ou à leur reconnaissance, tandis que la fonction « phrases » est considérée comme une fonction supérieure. Cette fonction est liée à la compréhension du langage et à la mémoire. Ainsi, toutes ces différences cognitives doivent être reflétées topographiquement. La Figure 4 permet de visualiser les données/coordonnées extraites à partir de la littérature correspondant aux fonctions « parole » et « phrases » (colorées respectivement en violet et magenta). On remarque la forme en « V » qui caractérise ces deux fonctions. Malgré la proximité de ces fonctions, leurs topographies sont clairement différentes.
De même, dans le graphe correspondant, nous remarquons qu’au niveau des interactions avec les autres fonctions cognitives, ce graphe est organisé en deux ensembles. Le premier est lié aux fonctions « parole » (connecté au nœud violet) et constitue les fonctions qui se caractérisent par une topographie similaire à celle de la « parole », telles que les voyelles, la production de syllabes, etc.
Le second ensemble est lié à la fonction « phrases » (connecté au nœud magenta) et représente les fonctions cognitives qui se chevauchent topographiquement avec les « phrases », telles que la compréhension de la parole, le traitement du langage, etc.
Documentation et ajout de connaissances autour de nouvelles données expérimentales
Grâce aux différentes bases de données intégrées dans LinkRbrain et à son concept novateur basé sur le calcul de similarités, il est possible, en quelques clics, de télécharger ces données sous forme d’images IRM ou de listes de coordonnées MNI dans le logiciel. LinkRbrain permet par la suite de visualiser :
- les données cartographiées sur un cerveau en 2D et 3D ;
- un graphe de connaissances en lien avec les mêmes régions topographiques ;
- une liste ordonnée de connaissances (cognitives ou génétiques) en lien avec les données ;
- une liste de références en lien avec les données.
LinkRfiber permet quant à lui d’accéder à l’ensemble des autres régions du cerveau qui sont connectées par des fibres avec les régions étudiées.
Génération d’hypothèses pour un problème multi-échelle
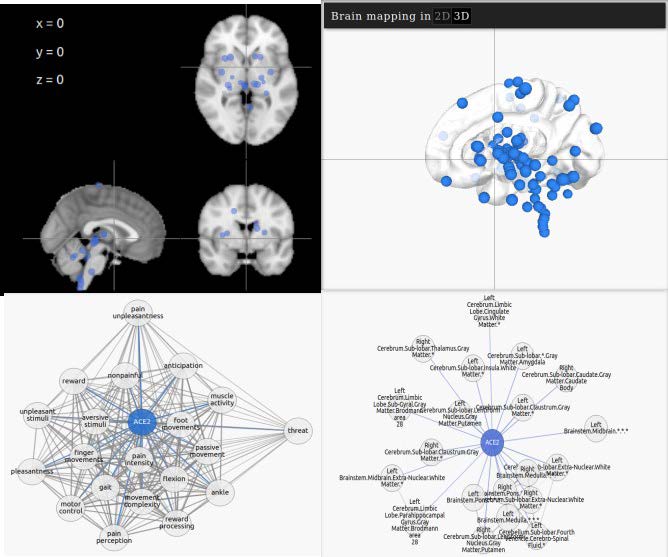
Pour caractériser l’étendue de l’impact complexe potentiel d’une infection par le SRAS-CoV-2 dans le cerveau, j’ai identifié quelles fonctions cognitives et sensorimotrices sont associées aux régions du cerveau où les récepteurs génétiques nommés Angiotensin Converting Enzyme 2 (ACE2) et Transmembrane Serine Protease 2 (TMPRSS2) sont sur-exprimés, en posant l’hypothèse qu’elles pourraient être particulièrement affectées. Les données sur les niveaux d’expression de ces gènes ont été fournies par l’Allen Institute for Brain Science, et la localisation des fonctions cérébrales par la plateforme LinkRbrain. Cette dernière a également été utilisée pour analyser le chevauchement spatial entre la sur-expression de ACE2/TMPRSS2 et les activations cérébrales spécifiques à une fonction, ou la sur-expression régionale d’autres gènes. Les fonctions cognitives et sensorimotrices dont le rôle pourrait être altéré ont été classées en six groupes : mémoire et souvenir, fonction motrice et sensorielle, douleur, lucidité, émotion, récompense. De plus, j’ai catégorisé les gènes qui s’expriment de manière différentielle dans les mêmes régions où les niveaux d’expression ACE2/TMPRSS2 sont les plus élevés. Les pathologies et fonctions cellulaires dans lesquelles ces gènes sont reliés sont les maladies neurodégénératives, l’immunité, l’inflammation, le récepteur olfactif, le cancer/apoptose, la fonction exécutive, les sens, l’ischémie, la fonction motrice, la myélinisation, et la dépendance. Grâce à LinkRbrain, de nouvelles hypothèses ont été générées pour explorer les manifestations neurologiques conséquentes du Covid-19.
Perspectives
Grâce au financement reçu dans le cadre du programme de prématuration du CNRS, l’infrastructure de LinkRdata est devenue modulaire et réutilisable. Ainsi, il est plus facile de la déployer vers d’autres projets qui intéressent la recherche interdisciplinaire.
LinkRdata se projette vers une plateforme de la recherche et de transfert de technologie complètement dématérialisée. Il s’agit d’une plateforme basée sur une stratégie évolutive et intégrative dotée de son propre écosystème informatique dédié :
- au stockage ou partage (quand c’est possible) des données multi-échelles : il s’agit de fournir une solution de sauvegarde intégrée afin de garantir sa pérennité et sa sécurité ;
- à la mise à disposition des logiciels scientifiques ouverts et des infrastructures de calcul ;
- à l’auto-configuration du meilleur workflow possible en tant que service à partir de la spécification du meilleur modèle intégré possible ;
- à la mise à disposition d’accélérateur de connaissances intégrées par la diffusion de nouveaux modèles intégrés pour la recherche, l’ingénierie, l’éducation et les services en ligne sociétaux innovants générant de nouvelles opportunités d’emploi dans l’économie numérique ;
- à la mise à disposition d’un environnement éducatif exceptionnel pour les travaux expérimentaux et théoriques des étudiants de master et de doctorat ;
- au transfert des connaissances et des technologies de la recherche pour une utilisation citoyenne via des interfaces faciles et fiables.
LinkRbrain aide à combler le fossé entre la science et l’ingénierie, en associant dans la même plateforme des ingénieurs et des chercheurs et chercheuses pour l’observation intégrée de données humaines in silico et in vivo. Cette association interdisciplinaire fournira des modèles intégrés pour les pathologies du cerveau à haute valeur ajoutée pour la science, l’éducation et la société.
Salma Mesmoudi, ingénieur de recherche, Centre européen de sociologie et de sciences politiques (CESSP), responsable scientifique du projet LinkRdata